近期在深圳大鹏湾出现的鲸鱼“小布”成为各界关注的焦点。自6月29日以来,这头被称为“小布”的鲸鱼在大鹏湾快乐生活,已经成为新晋“网红”。近日,在海洋研究人员的科研攻关下,“小布”的真实身份信息终于揭开了。原来“小布”是一头小布氏鲸,值得注意的是小布氏鲸并不是小的布氏鲸,而是区别于布氏鲸的不同物种。
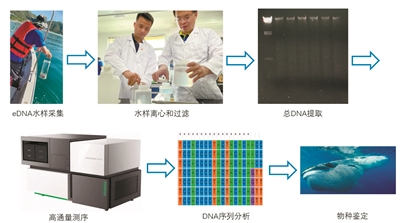
记者从华大海洋获悉,由华大海洋党支部书记、华大海洋研究院常务副院长游欣欣博士带领的科研攻关小组,运用环境DNA(eDNA)技术和高通量测序技术,从水体样本中初步鉴定出“小布”是小布氏鲸,这是全球首次利用环境DNA(eDNA)技术和高通量测序技术从水体样本中鉴定出这一物种。
文、图/广州日报全媒体记者王纳、轩慧
揭开“小布”身份之谜
据了解,自6月底“小布”惊现大鹏湾海域,华大海洋第一时间组建了以游欣欣博士为组长的科研攻关小组。攻关小组从“小布”活动的大鹏湾海域采集海水,经过连续多天攻坚,终于知道了这位“鲸”喜来宾的真实身份。
从水体中捕获到的DNA片段告诉我们,小布其实是一头小布氏鲸。小布氏鲸并不是小的布氏鲸,而是跟布氏鲸不同的物种。这是全球首次利用环境DNA(eDNA)技术和高通量测序技术从水体样本中鉴定到这一物种,并将其从一类很难通过形态学或声学进行区分的近源物种中分辨出来。
据悉,须鲸属是一类大型海洋哺乳动物,包含9个现生种,分别是4个曾被笼统划为布氏鲸的物种、普通小须鲸、南极小须鲸、长须鲸、黑板须鲸以及地球上有史以来最大的动物——蓝鲸。
那么,“小布”到底属于哪一种呢?华大海洋科研人员通过对“小布”所生活的水域进行eDNA水体样品采集及高通量测序,以四种布氏鲸的线粒体全基因组序列为参考序列,从5.2×109条测序序列中成功捕获到6条能比对到参考序列的序列(reads)。
这6条reads组装出4条长度约150bp~240bp不等的小布线粒体基因序列,它们分别位于tRNA-Met、ND2、ND4及CYTB区。这4条序列与保存在日本东京国立科学博物馆里的小布氏鲸样本的序列相似性分别为99%、100%、100%和100%,而与其他三个物种的序列相似性只有95%~97%。
因此,可初步确定“小布”是一种小布氏鲸。
只取一瓢水 便可知物种
究竟是什么样的技能,让我们“取一瓢水”而知天下?
记者从华大海洋了解到,其实,eDNA是指在环境样品中所有被发现的不同生物的基因组DNA的混合,生物脱落的表皮细胞或粪便是eDNA的主要来源。通过环境样品采集、过滤eDNA样品中的杂质,收集、浓缩和提取样品中的DNA,再通过PCR和高通量测序技术放大、解读这些DNA序列,理论上就可以知道环境中都有哪些物种了。
通常,eDNA技术主要用于检测陆生和水体环境中的微生物(细菌、真菌),近些年开始在水生脊椎动物研究中崭露头角,例如无刺蝠鲼、鲨鱼等软骨鱼类和多种硬骨鱼类。而针对个体较大的海洋哺乳动物的研究却鲜有报道,目前国际上尚无采用eDNA技术鉴定小布氏鲸这一类物种的报道。
相较于传统的研究方法而言,新兴的eDNA技术有很多优势。传统的大型海洋生物研究方法有组织取样、声学调查、卫星标记、航测和照片识别等等,但这些方法仅能在天气条件好、能见度高的时候适用于活跃于水体表层的个体,限制因素繁多。
水体eDNA技术不仅能克服以上种种限制因素,还能极大地提高检测灵敏度,最重要的是,它对所研究的生物不造成任何影响和伤害。
但是,在茫茫大海捕获eDNA,难度非常大,海洋中的DNA受水流、温度以及酸碱性等影响,在较短的时间内被稀释甚至降解。为了不惊扰到“小布”,采集水样的点需要远离“小布”的出没点,这样捕获到“小布”eDNA的概率大大降低。可以说,成功的概率很低。
据介绍,在开展此项工作的初期,华大海洋科研人员采用传统的方法检测“小布”eDNA,结果一无所获。后续,他们大胆尝试了新思路,采用了新技术,终于捕获到小布的eDNA,为大家揭开了“小布”这位“神秘来客”的真实身份。
未来,华大海洋会进一步对获得的eDNA数据进行分析,解读数据中鱼类的DNA信息,评估“小布”在大鹏湾活动海域的鱼类种类及其丰度,推测“小布”的饮食结构,为更好地“招待”这位“大朋友”提供参考借鉴,为大型鲸豚类的保护提供科学依据。